研究某个基因功能,往往从获得基因功能(gain of function)和缺失基因功能(loss of function)两个方向设计实验。目前CRISPR、RNAi干扰技术是缺失基因功能探究的主要手段,与CRISPR基因敲除不同,RNAi干扰技术不改变靶细胞的基因组序列,通过在转录水平上特异性打靶目的基因的一条或多条mRNA调节基因的表达。因此,该技术广泛应用于基因功能研究、肿瘤及病毒感染性疾病治疗等方面。
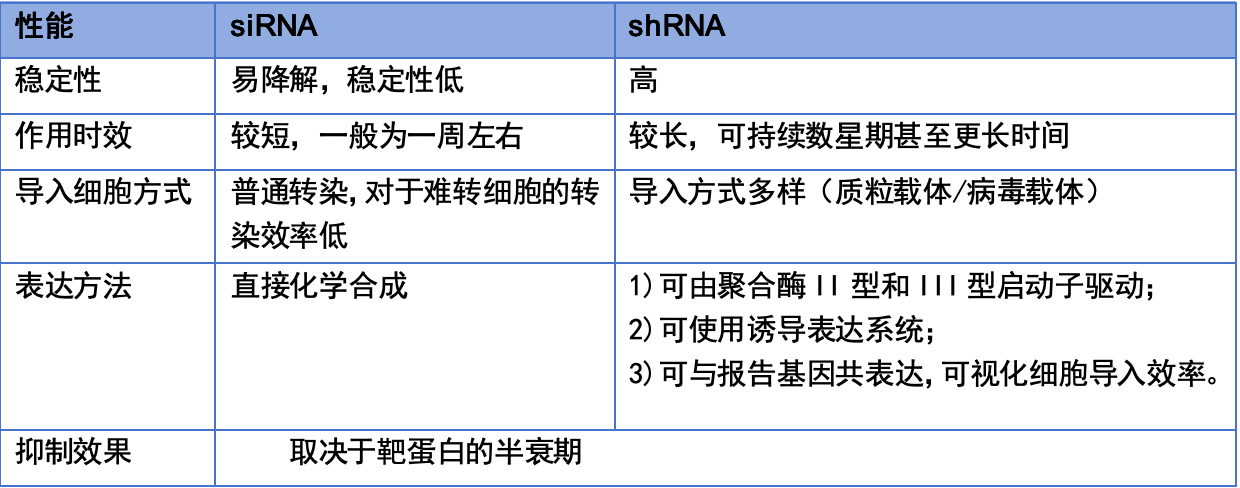
在进行基因敲低的实验过程中,通常会使用两种方式:化学合成的小干扰RNA(small interfering RNA,siRNA)和基于载体的的短发夹RNA(short hairpin RNA,shRNA)。这两种RNA分子都能与目标基因的mRNA分子特异性结合,并引导其降解,从而降低目的基因的表达水平。siRNA是一种双链RNA分子,当siRNA被导入细胞后,它会与细胞质中特定的酶形成 RNA 诱导沉默复合物(RNA-induced silencing complex,RISC),在 RISC的作用下,siRNA 解链成为单链,其中反义链特异识别并结合目的基因mRNA,随后对其进行剪切,通过诱导mRNA降解,降低蛋白翻译水平,从而调控目的基因的表达。而shRNA则是一种能够形成发夹结构的RNA分子,它可以在细胞内被加工成siRNA,进而发挥RNAi作用。
接下来看看具体的实验操作流程是怎样的吧!
根据实验研究内容确定需要沉默表达的目的基因。
一个基因可能存在多个名称缩写,为方便基因信息的统一,NCBI网站中每个基因都具有唯一的Gene ID编号。目的基因的转录本信息以及CDS序列均可在NCBI网站查询(网站链接:https://www.ncbi.nlm.nih.gov/gene)。
根据目标基因的序列,设计并合成能与目标基因特异性结合的siRNA或者shRNA。前者通过直接合成靶向目标基因的siRNA序列(19-27bp),将siRNA转染到细胞中进行表达。后者是通过将siRNA转化为shRNA(编码siRNA正义链和反义链,并且之间由一段loop序列隔开),构建表达shRNA的载体,以质粒或病毒为工具在细胞、活体中表达。
所以在设计RNA分子之前可根据实验目的选择siRNA或者shRNA,二者主要区别如下:
shRNA的设计可利用相关设计网站设计,例如Sigma、Life Technologies、GPP等网站。其中Sigma网站提供了一个较为全面的针对人和小鼠的shRNA库,并且部分基因的shRNA序列经过验证,可以直接使用,方便快捷。以人源TP53基因为例:
1.进入网站:https://www.sigmaaldrich.cn/CN/zh/semi-configurators/shrna?activeLink=productSearch 输入基因名称或Gene ID,点击查找。
|
|
2.选择物种-人源-确校
 |
3.查看序列信息(网站中部分基因的shRNA序列经过验证,可以选择验证过且敲低效率较高的序列)。
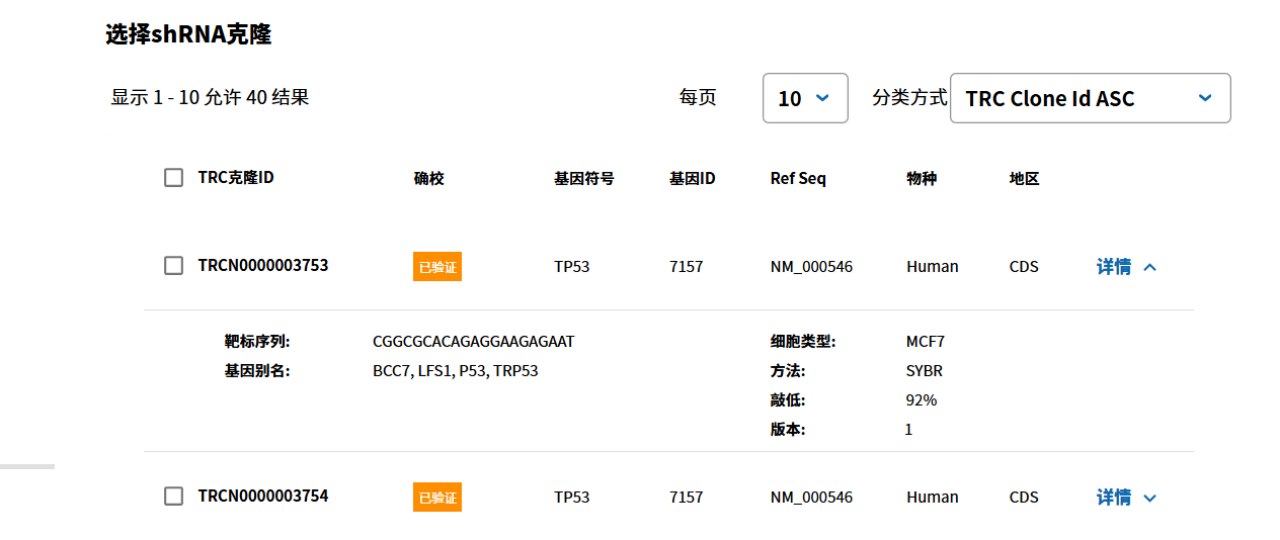 |
4.选择网站中靶点序列在NCBI网站中与目标基因进行BLAST,NCBI网址链接:https://blast.ncbi.nlm.nih.gov/Blast.cgi 注意设计的shRNA序列需特异性靶向目标基因,且与其它基因错配碱基须大于3个。
若通过直接转染siRNA进行基因沉默,直接进行化学合成即可。若通过质粒或病毒如慢病毒(LV)、腺相关病毒(AAV)长期或体内递送siRNA,需提前构建合适的核心质粒,并生产相应的质粒或病毒颗粒使其在细胞或体内进行表达。选择合适的表达载体至关重要,以AAV为例:
1.基于U6/H1启动子的 shRNA 干扰载体
shRNA是一类小/短发夹的双链RNA,由于构建序列较短,需使用特定的pol III型启动子U6或者H1实现体内的表达。U6/H1启动子具有表达能力强,表达范围广等优点,常用于实现全脑区或组织内的基因干扰。以AAV病毒核心质粒为例,载体结构如下:
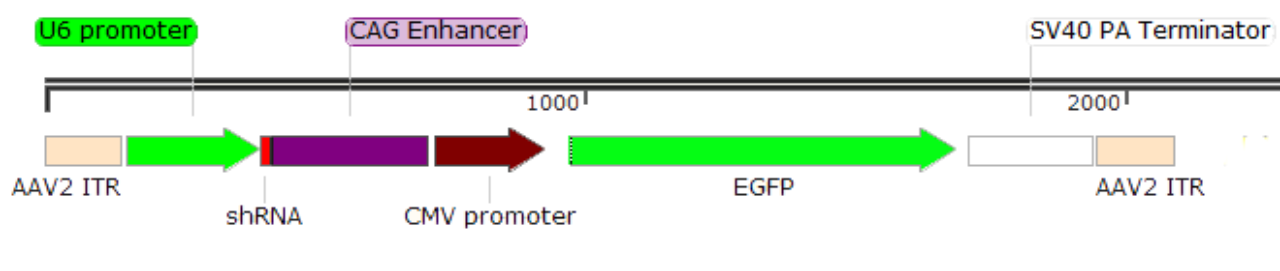
AAV-U6-shRNA表达载体结构示例
2.基于miR30骨架的 shRNA 干扰载体
在多数实验研究中,研究者需要实现在某组织或细胞内特异性敲低,而U6和H1不具有组织特异性,若想实现特定组织中的基因沉默,则需使用组织特异性启动子。研究人员利用miRNA和shRNA相似的合成机制,使用pre-miRNA作为生产siRNA的骨架,使得插入miRNA骨架的shRNA序列更长,因此可由pol II型启动子(包括多种组织特异性启动子,例如hSyn、GFAP)驱动,从而实现特定组织中的基因沉默[1] 。目前用于基因干扰的miRNA骨架有miR30、miR155、miR17-92等,其中最常用的就是miR30结构。以AAV病毒核心质粒为例,载体结构如下:
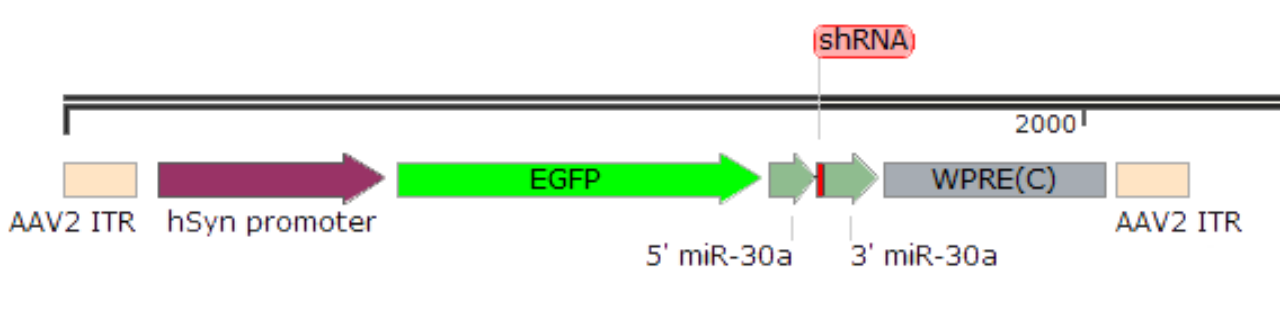
AAV-miR30-shRNA表达载体结构示例
3. miR30-shRNA载体结构的优势:
(1)通过启动子实现特异性。可根据不同II型启动子的特性实现特定组织或细胞的特异性表达。包括不局限于广谱启动子CMV、CAG、EF1α及组织特异性启动子hSyn等。
(2)实现多条shRNA共表达。基于miR30骨架研发出可同时表达2条shRNA的载体(一个基因两个靶点或靶向两个基因),且前后2条shRNA的表达量基本无差异。
(3)保留的miR30的天然环状结构能确保精确的Dicer切割并减少脱靶效应。
(4)实现Cre、Flp依赖表达。miR30骨架载体中可插入重组酶loxP位点,与Cre病毒或Cre小鼠一起使用,具有表达特异和受Cre表达调控的优势。
将设计好的shRNA或siRNA通过转染或其他方式转入到细胞中。在细胞内,shRNA会被转录和表达,进而形成siRNA。待其表达一段时间后,可以提取表达的RNA、蛋白,通过实时荧光定量PCR(qRT-PCR)、Western Blot、免疫组化等技术检测目的基因在RNA及蛋白层面的表达水平,评估目的基因的敲低效果,确保成功抑制目的基因的表达。
布林凯斯除了可定制常规基因的干扰病毒,还可进行circRNA、IncRNA、microRNA等非编码 mRNA的干扰病毒定制。定制相关病毒可提供shRNA设计、shRNA载体构建以及shRNA在体外的干扰效率验证。
问:NCBI里查到的目的基因有3条mRNA,但是合成siRNA只能选择其中一条mRNA , 目前已有的文献也没有做这个的,该如何选择呢?
答:3条mRNA可能至少产生3个蛋白,研究哪个蛋白就选和这个蛋白相关的mRNA,如果不产生蛋白,就根据独立的功能对应的mRNA来研究,如果是研究这个基因的全部功能,可以设计各个mRNA对应的siRNA来分别研究。
问:阴性对照应该是与目的基因的序列无同源性的,那如何评价转染效率?
答:阴性对照可通过携带荧光蛋白标签用于检测,转染进细胞即可出现荧光,故可用于优化转染条件和评估转染效率。而干扰效率则和shRNA的设计相关。
问:请问细胞水平实验,选择载体表达的shRNA还是小片段的siRNA?请问两种在转染效率和沉默时间等有什么区别呢?
答:根据实验需要沉默的时间来确定是用shRNA还是siRNA,1周以内的短期抑制,采用siRNA较好,无需构建载体,节省时间,还可以采用多次转染的方法,延长RNAi作用的时间到2周。若需要2周以上长效敲低目的基因,采用shRNA表达载体较好,持续表达时间长,缺点则是需要构建相关载体。效率上,主要与shRNA的设计有关,且根据细胞种类、实验情况的不同而不同。
问:刚开始做实验,准备把RNAi的片段转入细胞内稳定表达,不知道是shRNA还是siRNA哪个比较好而且效率比较高?
答:稳定转染当然是shRNA。若细胞本身转染效率较低,还可将质粒包装成慢病毒进行细胞感染,提高感染效率。
问:一个载体里同时放两个针对不同基因的shRNA会不会相互影响?
答:关于一个载体是否可以同时表达2个shRNA,涉及到2个shRNA的表达和加工的问题,关键还是2个shRNA都需要顺利表达,这主要和表达载体及其表达效率有关系。至于蛋白之间的相互影响,主要看干扰的两个蛋白之间本身是否存在影响。
问:RNA干扰如果用慢病毒做稳转的细胞株,什么样的目的基因都可以用来干扰来做稳转的细胞株吗?若能稳转,这个筛选的过程大概是多长时间呢?
答:当然不是任何基因都适合建立稳转的细胞株,这里面有两个前提,一是细胞能稳定存活传代,二是目的基因的mRNA需要被稳定的干扰掉。这两个前提必须同时存在才算成功。换句话说,如果某mRNA被干扰会导致细胞不能存活或无法稳定传代,自然无法建立这样的带RNAi的稳定细胞株。
问:准备做细胞内某基因RNA干扰前后的细胞生物学行为,是siRNA 还是shRNA 的效率好呢?
答:单就siRNA和shRNA干扰效率而言,没有区别;但是若考虑到转染效率,因为shRNA是以质粒或病毒为工具体外进行感染,可以通过筛选保证100%细胞都是KD的。而siRNA为提高转染效率而使用的助染试剂会具有一定的细胞毒性,会削弱或叠加实验结果,如果靶蛋白功能非常强大不可或缺,那么可能会有比较明显的差异,且siRNA成本较高。shRNA的缺点是,对于体内一个功能非常重要的基因可能有多种互补的途径,经过长期筛选,可能存在表型衰减甚至无表型的情况,但成本低廉,且可以得到稳定的模型。所以互有优劣,但通常首选是shRNA,siRNA多数用于应急或者互补的情况。
参考文献:
[1] Fellmann C, Hoffmann T, Sridhar V, Hopfgartner B, Muhar M, Roth M, Lai DY, Barbosa IA, Kwon JS, Guan Y, Sinha N, Zuber J. An optimized microRNA backbone for effective single-copy RNAi. Cell Rep. 2013 Dec 26;5(6):1704-13.